A l'occasion de la sortie de l'article de LinkR, nous revenons sur les avancées de LinkR, un an après la saison d'accélération de Data For Good.
LinkR et Data For Good - un an après
Introduction
En février 2024 débutait la douzième saison d’accélération de Data For Good. Pendant trois mois, des dizaines de bénévoles se sont mobilisés pour nous accompagner dans notre projet de création d’une plateforme de data science en santé, open source, low-code et collaborative.
Ces trois mois ont été très riches en échanges et en discussions, qui ont permis de donner un énorme coup de boost à LinkR. Ceci a permis une refonte importante de la plateforme, plus robuste, plus intuitive, et enrichie de nouveaux plugins.
Un an plus tard, ce travail collectif trouve une belle concrétisation : la publication de notre article dans l’International Journal of Medical Informatics.
Nous tenions à cette occasion remercier vivement les membres de l’association Data For Good et les contributeurs de la saison 12, et revenir sur les avancées de LinkR au cours de l’année passée.
Création d’un site web
Peu de temps après la fin de la saison 12 de Data For Good, avec l’appuis de bénévoles, nous avons créé le site web de LinkR.
Ce site héberge une documentation complète, pensée à la fois pour les cliniciens, sans besoin de compétences en programmation, et pour les data scientists ou développeurs.
Nous avons créé une section Articles, où nous publions régulièrement des contenus permettant de démocratiser la data science en santé : de l’explication de ce qu’est un entrepôt de données de santé à des guides plus techniques comme les requêtes SQL usuelles dans OMOP.
Enfin, une démo interactive permet de tester l’application en ligne, sans installation préalable.
Refonte de l’interface utilisateur
L’association InterHop est composée à la fois de cliniciens, de data scientists et de développeurs, ce qui a permis à LinkR d’évoluer grâce à une collaboration étroite entre expertises complémentaires.
Ce rapprochement direct entre utilisateurs finaux et développeurs a permis de concevoir rapidement une interface intuitive, adaptée aux pratiques concrètes en santé : exploration de données cliniques, création de tableaux de bord, construction de pipelines d’analyse, etc.
Intégration de Python
Si R reste un langage de référence pour l’analyse statistique, de nombreuses librairies incontournables de data science sont aujourd’hui disponibles uniquement en Python.
C’est pourquoi nous avons veillé à ce qu’il soit simple d’écrire et d’exécuter du code Python directement depuis l’interface de LinkR. Cette intégration élargit considérablement les possibilités de la plateforme, notamment pour le développement de plugins personnalisés s’appuyant sur des outils comme scikit-learn, pytorch, ou encore transformers.
Grâce à cette compatibilité, LinkR combine le meilleur des deux mondes : R et Python.
Création de nouveaux plugins
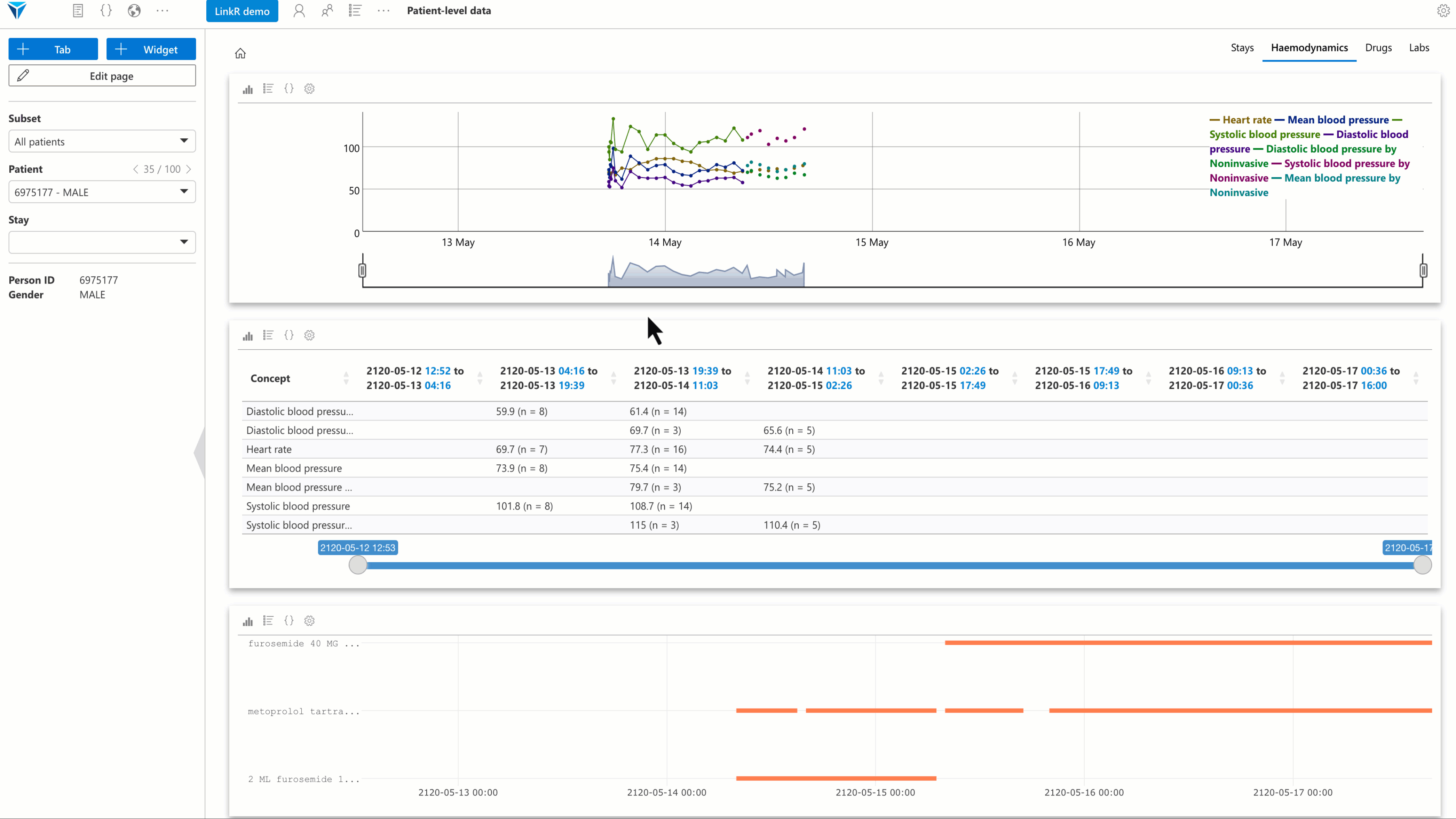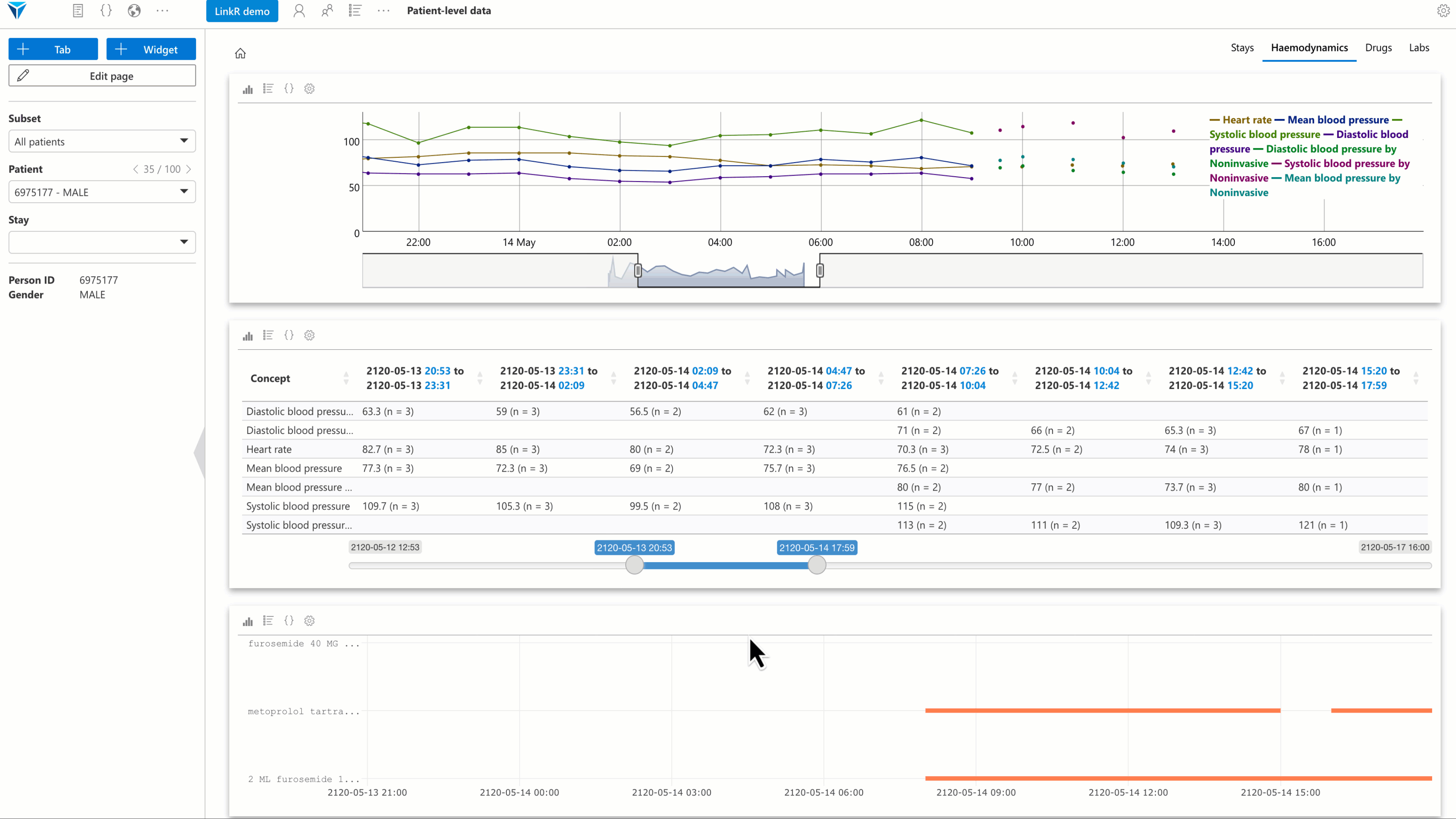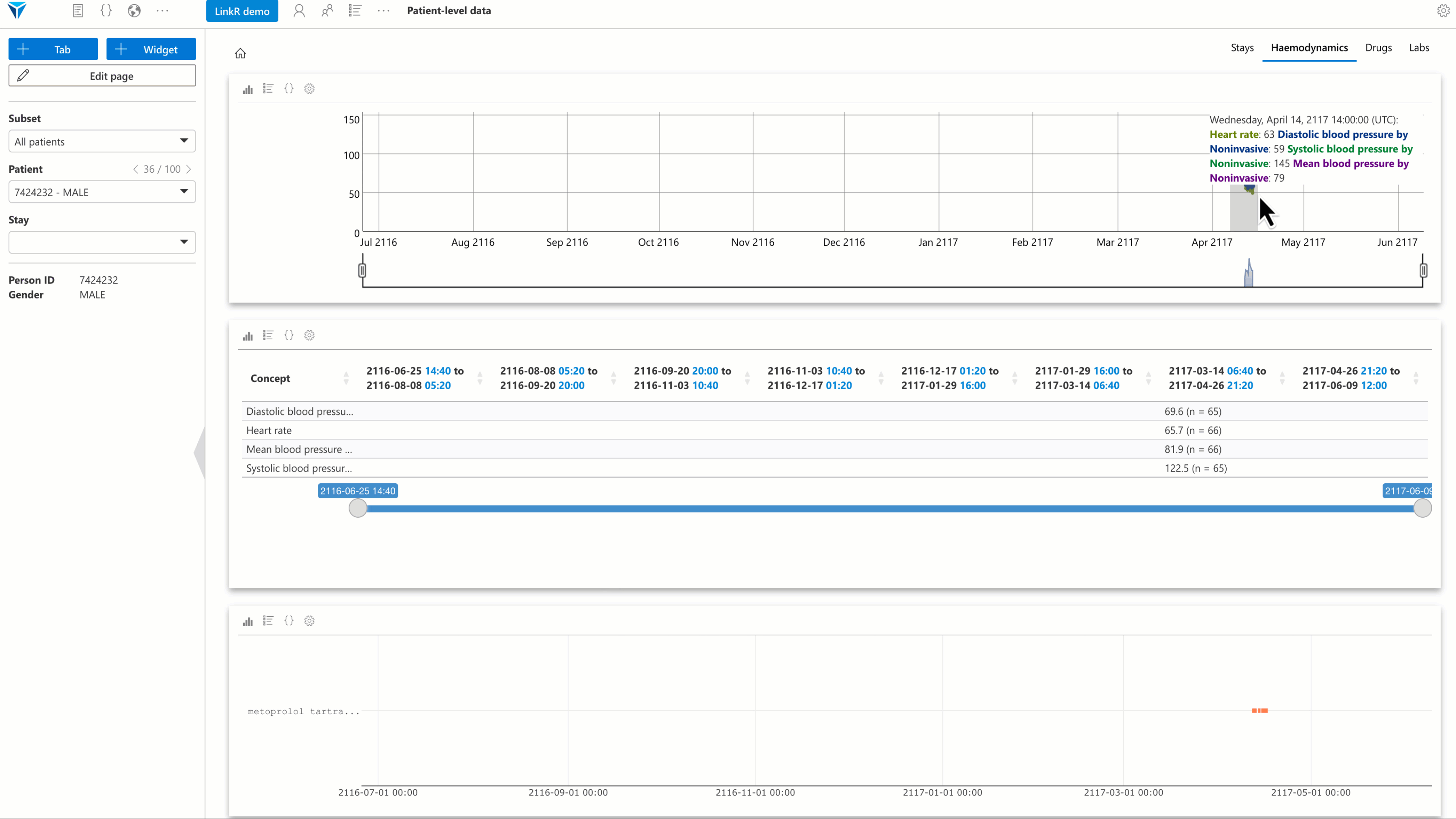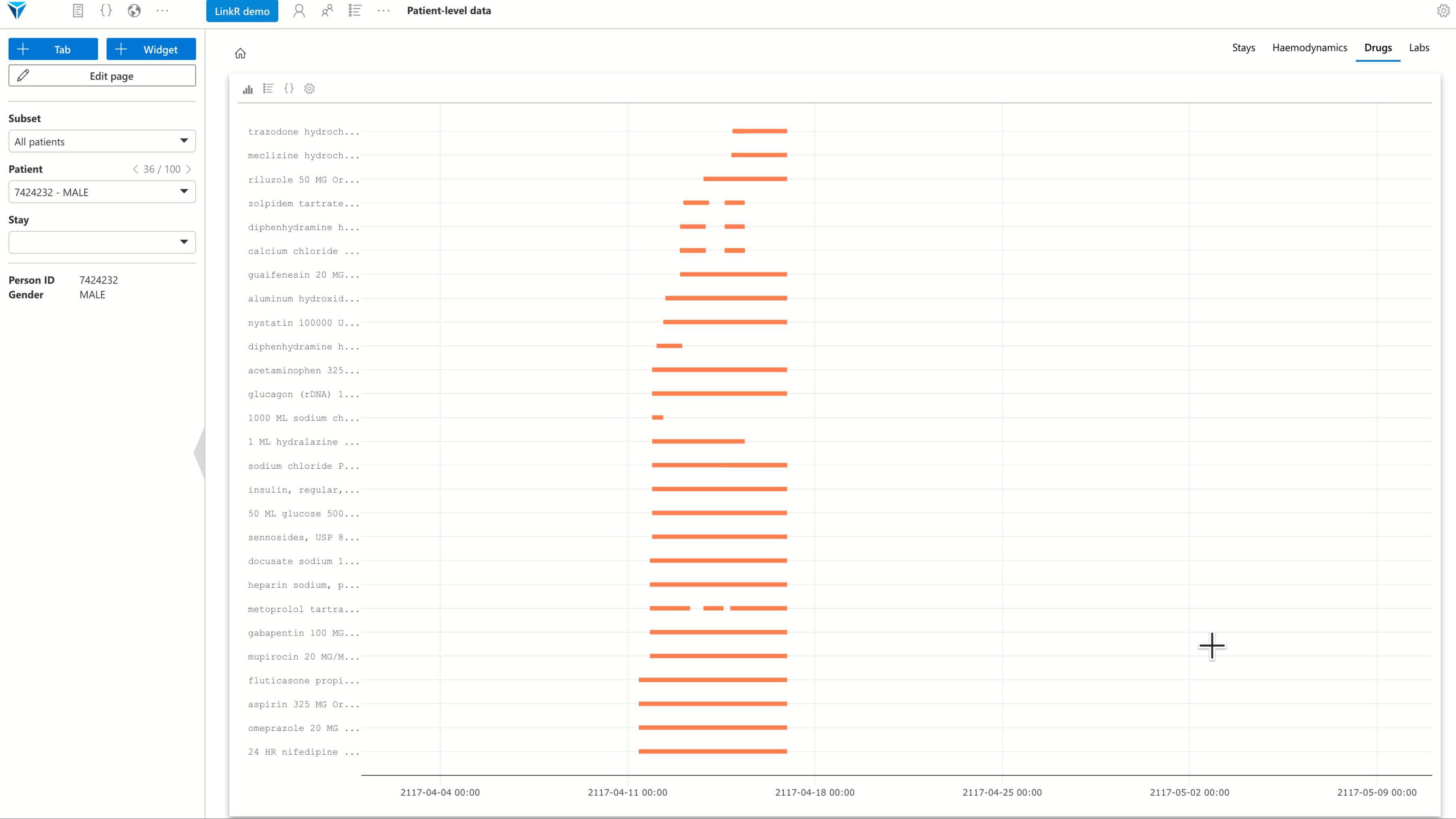
Plusieurs plugins ont été développés, avec un accent particulier sur l’analyse des données individuelles. L’objectif est de permettre à l’utilisateur de reconstituer, directement depuis l’environnement de recherche, une vue synthétique et interactive du dossier patient.
Voici quelques exemples de plugins disponibles :
- Plugin Timeline : permet de visualiser les données (paramètres vitaux, résultats biologiques, etc.) sous forme de frise chronologique interactive.
- Plugin Lecteur de texte : permet d’afficher les comptes rendus médicaux et d’effectuer des recherches dans le texte.
- Plugin Traitements reçus : permet de consulter les traitements administrés à un patient au fil du temps.
Comme toujours dans LinkR, chaque plugin dispose à la fois :
- d’une interface graphique pour visualiser les données et appliquer des filtres facilement
- d’une interface de programmation pour aller plus loin en modifiant ou étendant le code selon les besoins
Enfin, un plugin Template permet de guider les développeurs dans la création de nouveau plugins.
Utilisation de DuckDB
LinkR permet d’importer des données issues de bases relationnelles, mais aussi de fichiers CSV ou Parquet.
Pour garantir une interopérabilité maximale des analyses, nous avons fait le choix d’utiliser systématiquement des requêtes SQL comme langage commun.
Dans ce contexte, DuckDB s’est imposé comme une solution idéale : il permet de créer en quelques secondes des bases de données à partir de fichiers CSV ou Parquet, tout en offrant des performances remarquables. Grâce à lui, LinkR peut manipuler des tables de plusieurs millions de lignes, même sur des infrastructures limitées.
Quelle que soit la source des données, celles-ci peuvent être explorées et analysées en SQL, directement depuis l’interface de la plateforme.
Réflexion sur une v2
LinkR, dans sa version actuelle, est codé en R et repose principalement sur la librairie Shiny. Cette librairie, présentant l’avantage de faciliter et d’accélérer la création d’applications web, nous a permis d’obtenir rapidement un prototype fonctionnel.
Cependant, la communauté de développeurs codant avec Shiny en R reste très limitée, ce qui limite les possibilités de contribution sur notre projet.
Or, en tant que projet open source, nous avons besoin de la contribution de la communauté pour faire vivre le projet.
Ceci nous amène donc à mener une réflexion sur une nouvelle version de LinkR, qui s’appuiera sur des langages et librairies robustes et largement utilisés par la communauté de développeurs web.
Recherche de financements
InterHop est à la recherche de financements, afin de pouvoir débuter le développement de la v2.
Pour cela, nous allons candidater à la candidature de NLnet.
Si vous souhaitez nous aider, vous pouvez également faire un don à l’association.
Conclusion
Un an après Data For Good, LinkR a beaucoup évolué : refonte de l’interface, nouveaux plugins, intégration de Python, documentation enrichie, et publication scientifique.
Ce projet n’existerait pas sans l’engagement des contributeurs et contributrices qui ont permis d’en faire un outil accessible et open source.
Avec la v2 en préparation, l’aventure continue, et toutes les bonnes volontés sont les bienvenues pour y participer.
Datathon InterHop 2024
L’association InterHop organise en septembre 2024 un datathon dédié aux données de santé, mettant à disposition des participant·es la base de données MIMIC au format OMOP. Cet événement est une opportunité unique de collaborer autour de projets de data science, d’ingénierie des données et d’intelligence artificielle appliqués à la santé.
Modalités pratiques
Les participant·es sont invité·es à anticiper la création de leur compte sur PhysioNet, incluant la signature de l’accord d’utilisation des données (DUA, ou Data Use Agreement).
Un premier rendez-vous est prévu le jeudi 8 août à 13h00, dans le cadre d’une réunion interCHU pour constituer les équipes et affiner les projets.
Retrouvez toutes les informations sur les modalités pratiques du datathon dans cet article dédié.
Les thématiques et projets proposés
Durant ces 48 heures, plusieurs thèmes clés seront abordés, allant de la qualité des données à la prédiction de mortalité en passant par la visualisation d’indicateurs de santé.
1. FINESS+
Mettre à jour les données géographiques des établissements de santé pour garantir leur interopérabilité avec les systèmes ouverts.
- Thèmes principaux : Data engineering, Open data
- Objectif : Mettre à jour et structurer les données géographiques des 100 000 établissements de soins recensés au FINESS, afin de les rendre interopérables avec des systèmes ouverts comme OpenStreetMap.
- Intérêt : Permet de croiser les bases de données existantes et d’améliorer leur qualité, avec une contribution possible à Toobib.org.
2. Indicateurs d’activité de maternité
Développer des tableaux de bord interactifs pour analyser et suivre les indicateurs d’activité des maternités.
- Thèmes principaux : Data visualization, Data science
- Objectif : Créer des tableaux de bord dynamiques permettant de visualiser les indicateurs annuels d’activité des maternités (nombre de césariennes, transferts, péridurales, etc.).
- Méthodologie : Utilisation de LinkR, plateforme open-source facilitant l’analyse des données de santé.
- Impact attendu : Aide à l’amélioration du pilotage des services de maternité et au suivi des pratiques obstétricales.
3. Qualité des données
Détecter et corriger les biais dans les données de santé
- Thèmes principaux : Data cleaning, Pré-processing
- Objectif : Produire des indicateurs de qualité des données, permettant d’évaluer les biais potentiels liés aux logiciels de saisie et d’améliorer la fiabilité des données cliniques.
- Méthodologie : Comparaison de variables issues de la base MIMIC-OMOP avec des méthodes statistiques et des modèles de détection des anomalies.
- Impact attendu : Amélioration de la qualité des données de santé et facilitation de leur réutilisation pour la recherche.
4. Prédiction de la mortalité
Construire des modèles de prédiction de la mortalité pour améliorer la comparabilité des patients
- Thèmes principaux : Machine learning, Statistiques
- Objectif : Développer et comparer des modèles prédictifs de mortalité en réanimation (régression logistique, Random Forest, XGBoost, réseaux de neurones).
- Méthodologie : Entraînement de modèles sur MIMIC-OMOP et comparaison avec les scores traditionnels SOFA et IGS-2.
- Impact attendu : Création d’un modèle interopérable applicable sur des données locales pour améliorer la comparabilité des patients dans les études.
5. Aide au codage CIM-10
Aider le codage CIM-10 grâce à l’IA
- Thèmes principaux : NLP, Large Language Models
- Objectif : Automatiser l’identification des codes CIM-10.
- Méthodologie : Utilisation d’une approche basée sur les LLM et le RAG (Retrieval-Augmented Generation).
- Impact attendu : Amélioration de la précision et de l’efficacité du codage médical.
Conclusion
Ce datathon s’inscrit dans une démarche de science ouverte, avec une production de code interopérable et réutilisable pour l’ensemble de la communauté médicale et scientifique.
Chaque projet vise à améliorer la qualité, la visualisation et l’exploitation des données de santé, tout en facilitant la collaboration entre les cliniciens, data scientists et développeurs.
Le code source produit sera mis à disposition sur Framagit, et les résultats pourront être intégrés dans des plateformes comme LinkR pour être réutilisés et améliorés après l’événement.