Introduction
LinkR s’articule autour de projets, dans lesquels des sets de données sont chargés, contenant des données au format OMOP et utilisant des terminologies standards.
Ces données peuvent être mises en qualité grâce à l’utilisation et au partage de scripts de data cleaning.
Dans ces projets, l’utilisateur peut visualiser et analyser les données à l’aide de widgets, qui sont des plugins paramétrés.
Il est également possible d’accéder à un environnement de développement R et Python via la console.
Sets de données
LinkR fonctionne avec OMOP, un modèle de données commun international pour les données de santé.
Vous pouvez importer des données depuis différentes sources : une base de données, des fichiers Parquet ou des fichiers CSV.
Un même set de données peut être utilisé dans plusieurs projets.
Pour l’instant il est nécessaire de coder en R le script d’import des données. Une interface graphique sera codée dans une prochaine version.
Terminologies
Le modèle de données OMOP se base sur des terminologies standards, tells que :
- LOINC pour la biologie
- SNOMED pour les diagnostics
- RxNorm pour les médicaments
- etc…
Toutes ces terminologies sont disponibles sur Athena.
Ces terminologies doivent être importées dans LinkR, pour avoir les noms des concepts correspondants aux ID des concepts (colonnes concept_id des tables OMOP).
Scripts de data cleaning
Les données importées au format OMOP nécessitent souvent d’être mises en qualité à l’aide de scripts de data cleaning.
Un exemple classique est représenté par les données de poids et taille, qui, du fait de la façon dont sont créés les logiciels de soin, comporte souvent des données aberrantes, par exemple par inversion entre les champs poids et taille.
Des scripts pour exclure ce type de données sont souvent réalisés. LinkR permet de faciliter le partage de tels scripts, qui, du fait de l’utilisation du modèle de données commun OMOP, seront susceptibles de fonctionner sur les différents sets de données importés dans LinkR.
Quelques autres exemples de scripts :
- calcul de scores, tels que l’IGS-2 ou le score SOFA
- calcul de diurèse, en faisant la somme des différents paramètres (sonde urinaire, néphrostomie etc)
- etc…
Projets
Un projet est un environnement R et Python où seront analysées des données.
Un projet peut correspondre à une étude (par exemple une étude sur la prédiciton de la mortalité), mais également à des analyse de données hors étude, tels que la création de tableaux de bord (un tableau de bord permettant de visualiser l’activité d’un service hospitalier par exemple).
Lors de la création d’un projet, l’utilisateur choisit les données à utiliser, depuis les sets de données chargés dans l’application.
Le projet s’articulera autour de deux pages principales :
-
Page de données individuelles : ici, l’utilisateur pourra recréer l’équivalent d’un dossier clinique, en créant des onglets où il configurera des widgets, par exemple :
-
un onglet “Hémodynamique” où nous créerons des widgets permettant de visualiser la fréquence cardiaque, la pression artérielle et les traitements anti-hypertenseurs reçus par le patient)
-
un onglet “Notes” où nous afficherons toutes les documents textuels concernant le patient (compte-rendus hospitaliers, notes cliniques quotidiennes etc)
-
un onglet “Infectiologie” où nous afficherons toutes les données concernant l’infectiologie (prélèvements bactériologiques, antibioques reçus etc)
-
etc…
-
-
Page de données agrégées : ici, l’utilisateur créera de la même façon des onglets où il configurera des widgets. Il s’agira d’analyses sur un groupe de patients, par exemple :
-
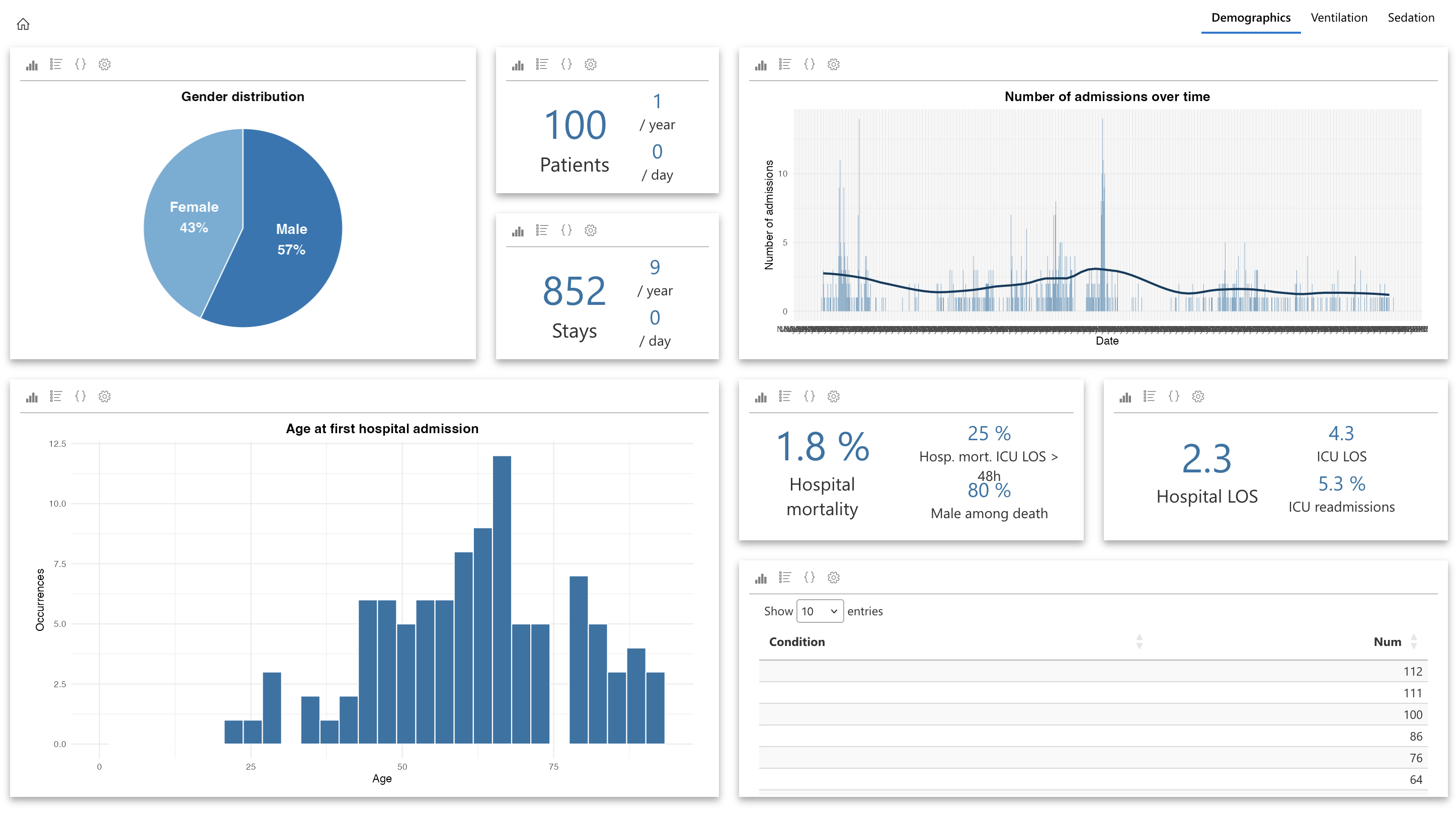
un onglet “Données démographiques” où l’utilisateur affichera les données démographiques du groupe de patients (âge, sexe, durée de séjour, mortalité etc)
-
un onglet “Données aberrantes” où sera affichée la distribution des différents paramètres et excluera les données aberrantes
-
un onglet “Analyse de survie” où un widget sera configuré pour réaliser l’analyse de survie de la population sélectionnée
-
etc…
-
A l’aide de l’interface low-code (qui associe une interface de code et une interface graphique), le travail collaboratif entre data scientists, statisticiens et cliniciens devient plus facile.
Voir comment créer un projet.
Plugins
Les plugins sont des briques de code R et Python qui permettent d’ajouter des fonctionnalités à LinkR.
Comme nous l’avons vu dans le paragraphe précédent, les projets s’articulent en onglets.
Ces onglets comportent des widgets, qui sont des plugins appliqués à des données.
Par exemple, si je choisir le plugin “Timeline” pour être appliqué au paramètre “Fréquence cardiaque”, le widget résultant sera un graphique sous forme de timeline qui affichera la fréquence cardiaque du patient sélectionné.
Il existe des plugins de données individuelles, qui sont les éléments permettant de recréer un dossier médical, par exemple :
- Lecteur de document : ce plugin permet d’afficher les documents textuels (compte-rendus hospitaliers, notes cliniques) et de les filtrer (avec recherche de mot clef ou avec filtre sur le titre par exemple)
- Timeline : comme évoqué ci-dessus, pour afficher les données temporelles sous forme de timeline
- Datatable : permet d’afficher les données sous forme de Datatable, comme par exemple l’affichage de la biologie d’un patient en fonction de l’horaire de prélèvement
- etc…
Nous avons aussi des plugins de données agrégées, qui serviront à visualiser et analyser des données agrégées, par exemple :
- ggplot2 : un plugin qui permet d’afficher les variables avec les différentes figures proposées par la librairie ggplot2
- Analyse de survie : permet de réaliser des analyses de survie
- Machine learning : pour entraîner et évaluer des modèles de machine learning, avec des librairies R ou Python
- etc…
Voir comment créer un plugin.
Widgets
Les widgets correspondent à des plugins appliqués à des données.
Après avoir créé un onglet, je peux y ajouter plusieurs widgets.
Ces widgets peuvent être changés de taille et déplacés sur la page.
Subsets
Au niveau d’un projet, un set de données peut être partagé en plusieurs subsets.
Un subset est un sous-ensemble du set de données global, après avoir appliqué des filtres pour sélectionner des patients.
Voici des exemples de subsets que l’on pourrait imaginer sur la base de données MIMIC, qui comporte les séjours de plus de 50 000 patients en réanimation, adulte et pédiatrique :
- Patients de plus de 18 ans admis en réanimation médicale pour COVID-19
- Patients ayant un code CIM-10 de pneumopathie infectieuse et ayant été traité par Amoxicilline
- Patients exclus : il peut être utile de créer un subset avec uniquement les patients exclus des analyses
- etc…
Pour l’instant il est nécessaire de coder en R les scripts pour créer les subsets. Une interface graphique sera codée dans une prochaine version.
Voir comment créer un subset.